基因的上下游分析

实验介绍基因的上下游分析是做生物医学研究必要的分析之一,但需要整合各种调控数据库以及多样本基因表达数据,某些研究人员可能无从下手!
您只需要提供感兴趣的基因(或者miRNA,TF)名称,云生信整合全世界权威公共数据库所有样本数据,从众多基因上下游的分析结果中,挑选出与疾病最相关的结果。结果作为实验的理论支持,大大降低实验的假阳性!
基因的上下游分析分析内容:
1. 转录因子调控靶基因分析(TF-target);
2. microRNA调控靶基因分析(miRNA-Target);
3. 蛋白质与蛋白质相互作用分析(PPI);
4. 其他,例如TF-miRNA、TF-lncRNA、lncRNA-mRNA、miRNA-lncRNA等。
云生信整合的全世界权威公共数据库所有样本数据:
1. 各个物种的转录调控数据库(TRANSFAC、JASPAR、TRED、PAZAR、AGRIS、RegulateDB、CHIPBASE等);
2. 各种miRNA调控靶基因的算法工具(miRBase、miRWalk、TargetScan、miRanda、PicTar、PITA等);
3. 蛋白互作数据库(如HPRD、STRING、DIP、BioGRID、MINT等);
4. TCGA数据库的RNA-seq、small-RNA-seq等;
5. GEO数据库的基因芯片、miRNAx芯片等。
图片可直接使用:
云生信团队的分析过程依托于云生信分析平台(www.bioclouservice.com ),获得的分析图片完全达到SCI论文的水平要求,可以直接使用。
分析耗时及报价:
基因的每种调控分析为1500元,与疾病关联的精确分析为2000元。多个基因或多种调控同时分析还有大优惠!
结直肠癌相关的miR-***的上游和下游调控关系分析样例
分析目的:
通过对结直肠癌公共芯片数据的分析,获得与结直肠癌相关的mir-***调控的上游、下游基因。
分析过程:
1. 公共芯片数据的选择下载。
本分析使用的数据下载自GEO(GSE4****)[1],样本来自1992年至2004年期间在MSK肿瘤中心接受诊治的结直肠癌患者,包括1**例结直肠癌样本,与5*例正常结直肠组织对照。芯片平台使用的是Affymetrix HG-U133A ,能够检测超过*****个人基因的转录表达水平。
2. 差异表达基因分析
我们使用R软件包Limma[2]中的经验贝叶斯模型识别1**例结直肠癌与5*例正常结直肠组织之间的差异表达基因,以logFC绝对值大于或者等于0.585(相当于1.5倍的平均表达水平改变)和adj.P.Val 小于0.05为基因显著差异表达的阈值。adj.P.Val是Benjamini & Hochberg方法[3]进行多重检验校正的结果。我们一共发现了522个显著上调基因和653个显著下调基因。
3. miRNA靶基因关系分析
使用miRecord数据库[4]的实验验证数据,以及miRecord数据库的预测数据。其中,miRecord预测工具我们使用了miranda[5],mirtarget2[6],pita[7],rnahybrid[8],targetscan[9]这5中预测方法进行分析,然后选择预测结果出现4次及以上的关系对作为 miRNA调控靶基因关系对。最后,我们将这些预测得到的靶基因与结直肠癌差异表达基因进行交集分析,得到了miRNA调控的差异表达基因信息。
miRecord Validated Targets数据库中,经过实验验证的miR-***调控靶基因只有一个结果,即 MTA1基因。miRecord Predicted Targets数据库中,选择大于等于4次的关系对,最终得到了2214个靶基因。
将实验验证结果+预测结果共计2215个靶基因与522个显著上调基因和653个显著下调基因取交集后,分别得到了49个上调靶基因和58个下调靶基因。如图1所示。
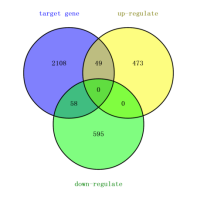
图1 miR-***的靶基因与差异表达基因的venn图
4. TF调控miRNA分析
我们使用CHIPbase[10]数据预测分析调控miR-***的转录因子(TF)信息,然后从中筛选出属于差异表达基因的TF。
利用CHIPbase数据库我们得到,调控miR-***的转录因子共有**个,其中属于结直肠癌差异表达基因的共有2个,即MYC,CEBPB。
5. 构建整合网络图。
结合miR-***的上游和下游分析结果,使用cytoscape[11]软件做图,结果如图2所示。
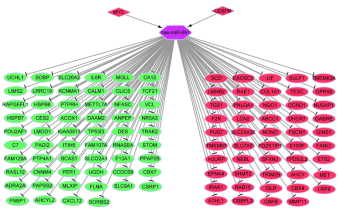
图2 整合网络图。菱形代表转录因子(TF);六边形代表miRNA;椭圆代表靶基因。红色代表差异上调基因,绿色代表差异下调基因。
结论
通过以上的分析,我们能够得到与疾病(结直肠癌)相关的差异表达基因列表,miR-***调控的靶基因以及被调控的转录因子,并从中获得与结直肠癌相关的miR-***的上游和下游关系。我们可以从中获得重要的转录因子,并挑选合适的靶基因。
参考文献1.Sheffer, M., et al., Association of survival and disease progression with chromosomal instability: a genomic exploration of colorectal cancer. Proceedings of the National Academy of Sciences of the United States of America, 2009. 106(17): p. 7131-6.
2.Smyth, G.K., Limma: linear models for microarray data, in Bioinformatics and Computational Biology Solutions Using {R} and Bioconductor, R.G.a.V.C.a.S.D.a.R.I.a.W. Huber, Editor. 2005, Springer: New York. p. 397--420.
3.Benjamini, Y.H., Yosef Controlling the false discovery rate: a practical and powerful approach to multiple testing. Journal of the Royal Statistical Society, Series B, 1995. 57(1): p. 289-300.
4.Xiao, F., et al., miRecords: an integrated resource for microRNA-target interactions. Nucleic acids research, 2009. 37(Database issue): p. D105-10.
5.John, B., et al., Human MicroRNA targets. PLoS biology, 2004. 2(11): p. e363.
6.Wang, X., miRDB: a microRNA target prediction and functional annotation database with a wiki interface. RNA, 2008. 14(6): p. 1012-7.
7.Kertesz, M., et al., The role of site accessibility in microRNA target recognition. Nature genetics, 2007. 39(10): p. 1278-84.
8.Kruger, J. and M. Rehmsmeier, RNAhybrid: microRNA target prediction easy, fast and flexible. Nucleic Acids Res, 2006. 34(Web Server issue): p. W451-4.
9.Agarwal, V., et al., Predicting effective microRNA target sites in mammalian mRNAs. Elife, 2015. 4.
10.Yang, J.H., et al., ChIPBase: a database for decoding the transcriptional regulation of long non-coding RNA and microRNA genes from ChIP-Seq data. Nucleic Acids Res, 2013. 41(Database issue): p. D177-87.
11.Smoot, M.E., et al., Cytoscape 2.8: new features for data integration and network visualization. Bioinformatics, 2011. 27(3): p. 431-432.




